https://habr.com/ru/company/skillfactory/blog/536592/- Блог компании SkillFactory
- Python
- Data Mining
- Big Data
- Data Engineering
В этой статье я хочу продемонстрировать R Markdown — удобную надстройку для программирования вашего проекта как на R, так и на Python, позволяющую программировать некоторые элементы вашего проекта на двух языках и управлять объектами, созданными на одном языке, с помощью другого языка. Это может быть полезно потому, что:
- Позволяет писать код на привычном языке, но при этом использовать функции, существующие только в другом языке.
- Позволяет напрямую сотрудничать с коллегой, который программирует на другом языке.
- Даёт возможность работать с двумя языками и со временем научиться свободно владеть ими.
Что нам понадобится
Для работы понадобятся эти компоненты:
- Конечно, R и Python.
- IDE RStudio (можно сделать это в других IDE, но в RStudio проще).
- Ваш любимый менеджер среды для Python (здесь я использую conda).
- Пакеты
rmarkdown и reticulate, установленные в R.
При написании документов R Markdown мы будем работать в среде RStudio, но при этом перемещаться между фрагментами кода, написанными на R и на Python. Покажу пару простых примеров.
Настройка среды Python
Если вы знакомы с программированием на Python, то вы знаете, что любая выполняемая на Python работа, должна ссылаться на конкретную, содержащую все необходимые для работы пакеты среду. Есть много способов управления пакетами в Python, два самых популярных — virtualenv и conda. Здесь я предполагаю, что мы используем conda и что он установлен в качестве менеджера среды Python.
Вы можете использовать пакет reticulate в R для настройки окружений conda через командную строку R, если хотите (используя такие функции, как
conda_create()), но как обычный программист Python я предпочитаю настраивать свои среды вручную.
Предположим, мы создаём среду conda с именем
r_and_python и устанавливаем в неё
pandas и
statsmodels. Итак, команды в терминале:
conda create -name r_and_python
conda activate r_and_python
conda install pandas
conda install statsmodels
После установки
pandas,
statsmodels (и любых других пакетов, которые могут вам понадобиться) настройка среды завершена. Теперь запустите conda info в терминале и выберите путь к вашей среде. Он понадобится вам на следующем шаге.
Настройка вашего проекта R для работы с R и Python
Мы запустим проект R в RStudio, но хотим иметь возможность запускать Python в этом же проекте. Чтобы убедиться, что код Python работает нужной нам среде, необходимо установить системную переменную среды
RETICULATE_PYTHON для исполняемого файла Python в этой среде. Это будет путь, который вы выбрали в предыдущем разделе, за которым следует
/bin/python3.
Лучший способ обеспечить постоянную установку этой переменной в вашем проекте — создать в проекте текстовый файл с именем
.Rprofile и добавить в него эту строку.
Sys.setenv(RETICULATE_PYTHON=”path_to_environment/bin/python3")
Замените pathtoenvironment на путь, который вы выбрали в предыдущем разделе. Сохраните файл
.Rprofile и перезапустите сеанс R. Каждый раз, когда вы перезапускаете сеанс или проект, запускается
.Rprofile, настраивающий вашу среду Python. Если вы хотите проверить это, вы можете запустить строку Sys.getenv («RETICULATE_PYTHON»).
Написание кода — первый пример
Теперь вы можете настроить в своём проекте документ R Markdown
.Rmd и писать код на двух разных языках. Сначала нужно загрузить библиотеку reticulate в ваш первый фрагмент кода.
```{r}
library(reticulate)
```
Теперь, когда вы захотите написать код на Python, можно обернуть его обычными обратными кавычками, но пометить как фрагмент кода Python с помощью
{python}, а когда захотите писать на R — воспользуйтесь
{r}.
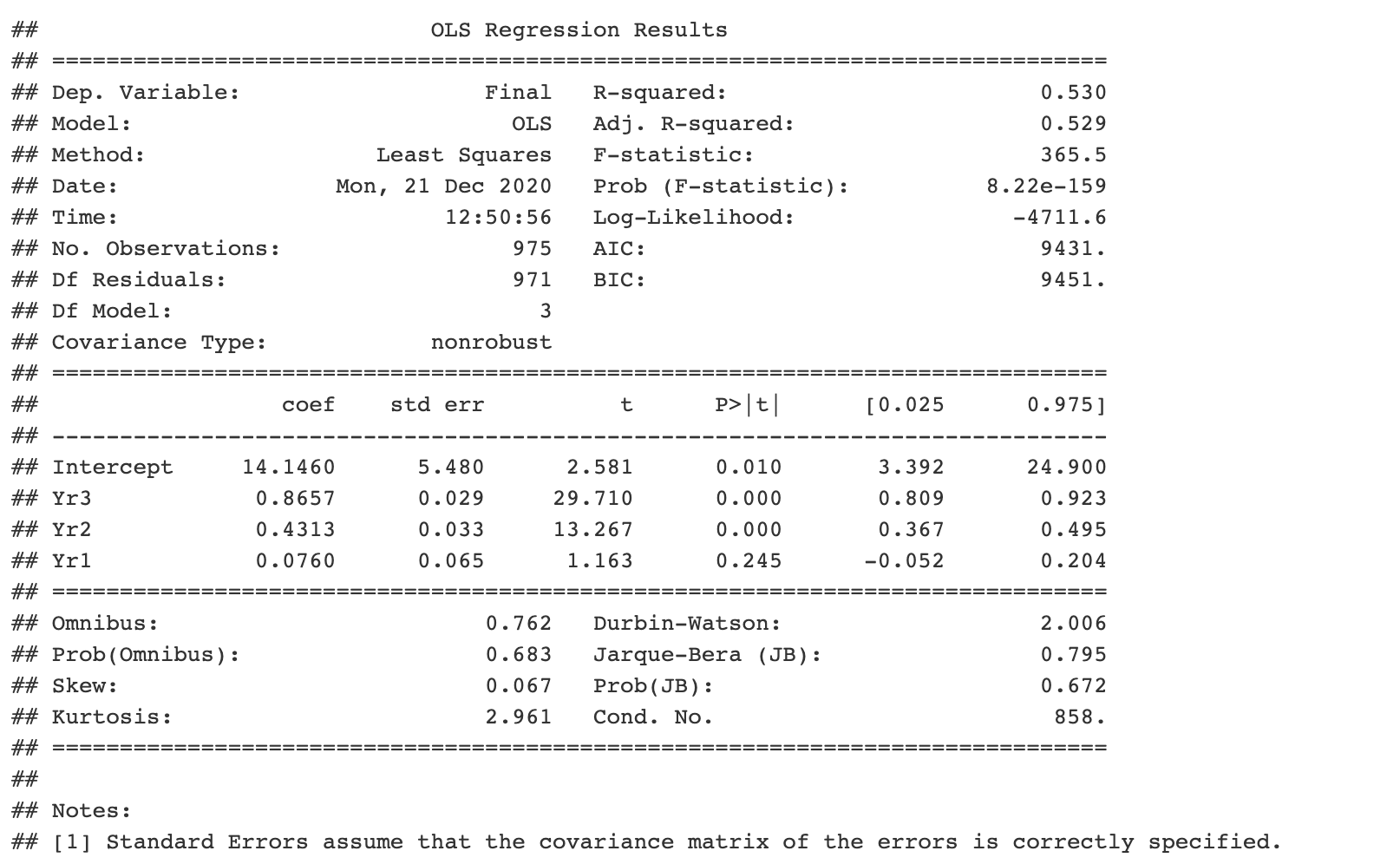
В нашем первом примере предположим, что вы запустили модель на Python на наборе данных с результатами тестов учащихся.
```{python}
import pandas as pd
import statsmodels.api as sm
import statsmodels.formula.api as smf
# obtain ugtests data
url = “http://peopleanalytics-regression-book.org/data/ugtests.csv"
ugtests = pd.read_csv(url)
# define model
model = smf.ols(formula = “Final ~ Yr3 + Yr2 + Yr1”, data = ugtests)
# fit model
fitted_model = model.fit()
# see results summary
model_summary = fitted_model.summary()
print(model_summary)
```
Это здорово, но, допустим, пришлось бросить работу из-за чего-то более срочного и передать ее своему коллеге, программисту на R. Вы надеялись, что сможете провести диагностику модели.
Не бойтесь. Можно получить доступ ко всем объектам python, которые вы создали в общем списке с названием py. Поэтому, если создается блок R внутри вашего документа R Markdown, коллеги получат доступ к параметрам вашей модели:
```{r}
py$fitted_model$params
```
или первые несколько остатков:
```{r}
py$fitted_model$resid[1:5]
```
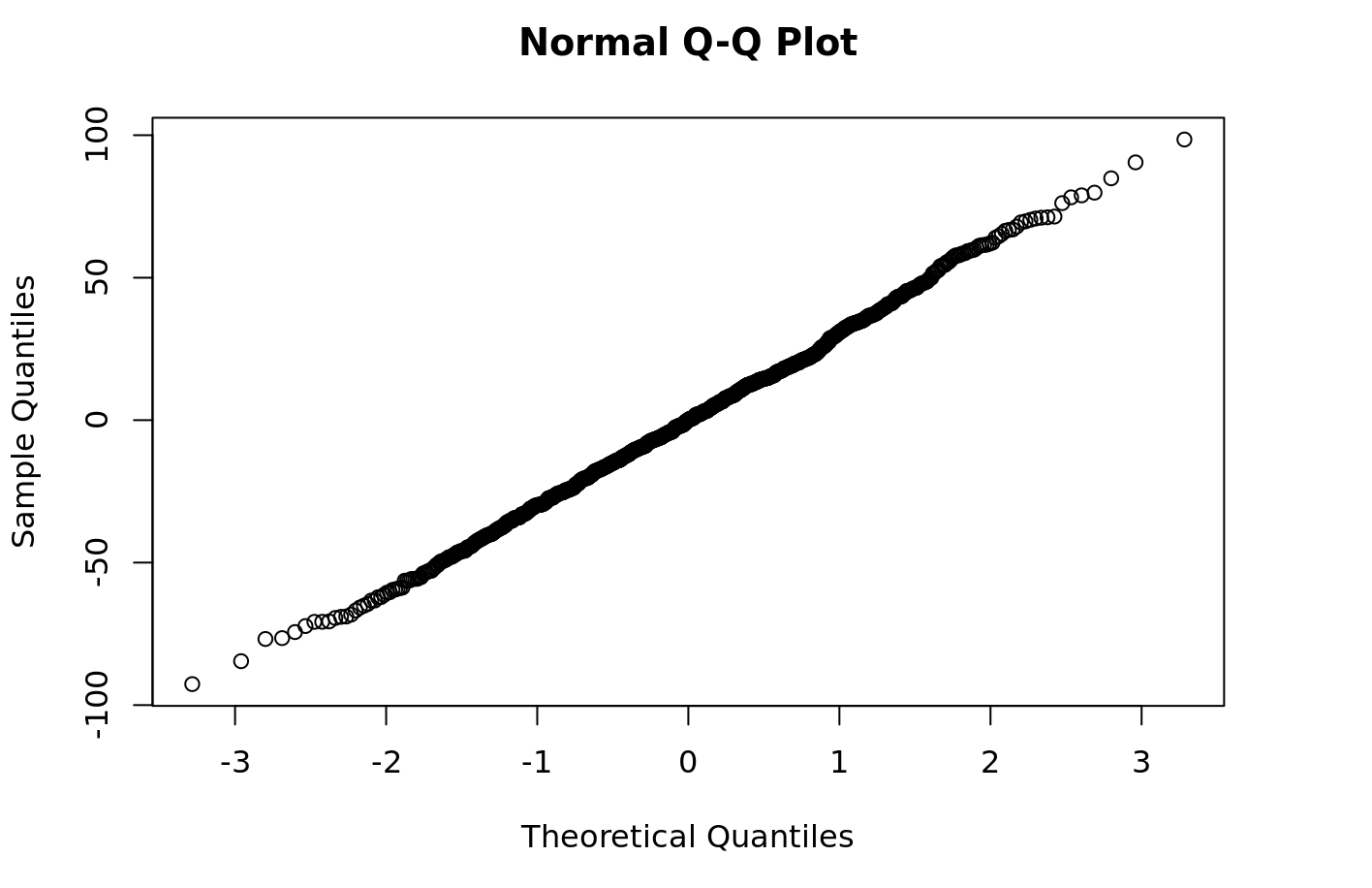
Теперь можно легко выполнить некоторую диагностику модели, например построить график остатков вашей модели типа квантиль-квантиль:
```{r}
qqnorm(py$fitted_model$resid)
```
Написание кода — второй пример
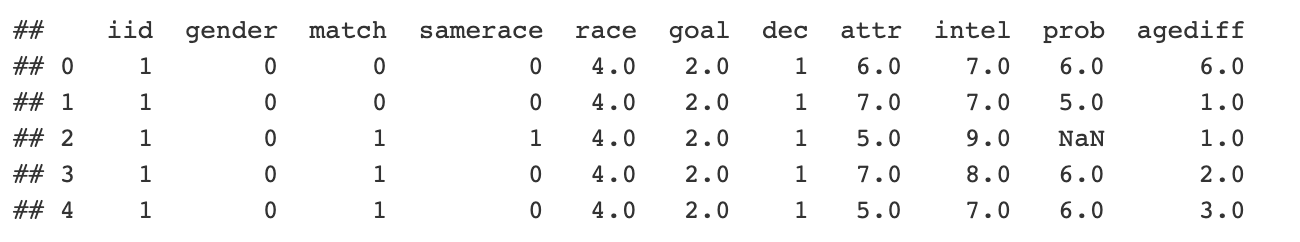
Вы анализировали некоторые данные о быстрых знакомствах на Python и создали фрейм данных pandas со всеми данными в нём. Для простоты загрузим данные и посмотрим на них:
```{python}
import pandas as pd
url = “http://peopleanalytics-regression-book.org/data/speed_dating.csv"
speed_dating = pd.read_csv(url)
print(speed_dating.head())
```
Теперь вы запустили простую модель логистической регрессии в Python, чтобы попытаться связать решение dec с некоторыми другими переменными. Однако вы понимаете, что эти данные на самом деле иерархичны и что один и тот же индивидуальный iid может иметь несколько знакомств.
Итак, вы понимаете, что вам нужно запустить модель логистической регрессии со смешанными эффектами, но не можете найти никакой программы на Python, которая бы сделала это!
И снова ничего не бойтесь, отправьте проект коллеге и он напишет решение на R.
```{r}
library(lme4)
speed_dating <- py$speed_dating
iid_intercept_model <- lme4:::glmer(dec ~ agediff + samerace + attr + intel + prob + (1 | iid),
data = speed_dating,
family = “binomial”)
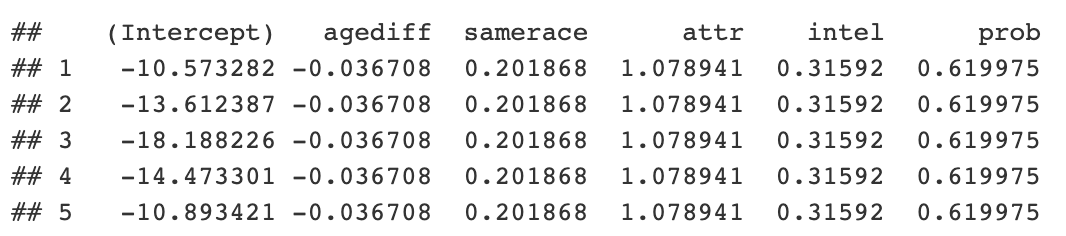
coefficients <- coef(iid_intercept_model)$iid
```
Теперь вы можете получить код и посмотреть на коэффициенты. Также можно получить доступ к объектам R в Python внутри общего объекта r.
```{python}
coefs = r.coefficients
print(coefs.head())
```
Эти два примера показывают, как можно плавно перемещаться между R и Python в одном документе R Markdown. Поэтому в следующий раз, когда вы подумаете о работе над межъязыковым проектом, подумайте и о том, чтобы запустить все его этапы в R Markdown. Это может избавить вас от множества проблем, связанных с переключением между двумя языками, и поможет сохранить всю вашу работу в одном месте, в виде непрерывного нарратива.
Вы можете увидеть готовый документ R Markdown, созданный на основе интеграции языков — с фрагментами R и Python и объектами, перемещающимися между ними, — опубликованный
здесь. Репозиторий Github с исходным кодом находится
здесь.
Примеры данных в документе взяты из моего
Справочника по моделированию регрессии в People Analytics.